今回は RNA velocity (直訳するとRNA速度だが、適切な語訳ではないため、RNA velocityとして記述する)について説明する。
RNA velocity の基礎知識
RNA velocity は2018年に La Manno らが発表・提唱したもので、(一般に)scRNA-seq (single cell RNA-seq; 単一細胞RNAシーケンス)における概念である。単一細胞の遺伝子発現の状態を経時的に追跡するというものではなく、その測定(シーケンス)の瞬間、つまり静的な状態からトランスクリプトーム空間における移動の方向と速度(ベクトル)を得る、というものだ。現状、シーケンスする際に細胞を殺してしまうため、瞬間的なデータ(スナップショット)しか得ることができないが、そこから細胞の状態、ひいては細胞の将来を予測しようというものである。
簡単に「トランスクリプトーム空間におけるRNAのベクトル」と言ってしまうが、ではどのようにそのベクトルを推定するのか。
mRNAはゲノムDNAからRNAポリメラーゼを介して転写される。一般的に真核生物においては転写される遺伝子領域内にイントロンを含むことが多い。実際、イントロンを含まない遺伝子は全体の遺伝子のうち2.3%であると見積もられている (Dvorak et al., 2023)。転写されたmRNA(pre-mRNA)はまだイントロンを含んでおり、未成熟な状態である。このあとスプライシングが起こり、成熟 mRNA となる。この「未成熟 mRNA」と「成熟 mRNA」は含まれるイントロン配列の有無によって区別することができ、それぞれの量によって「最近になって転写が活発になったか」「ダウンレギュレーションが起きているか」などがわかる。より一般化した言い方をすると、そのふたつの量の差分が遺伝子の転写の「変化量」を示すことになる。これにはRNAの分解速度などを含めた定常状態も考慮する必要がある。
下の図でいうところの微分方程式によって一つの遺伝子についての「速度(Velocity)」が求まる。pre-mRNA の成熟(スプライシング)には細胞ごとに時間差が生じるため、その時間差に基づいて遺伝子のRNA velocityのモデル化が可能となる(下図B)。
さらに、scRNA-seqによって大量の細胞ごとの転写量のデータ、および上記のようなモデルを元に高次元のトランスクリプトーム空間における細胞のベクトルを推定することができ、それに基づいて細胞の将来の状態を推定することも可能となる(下図C)。
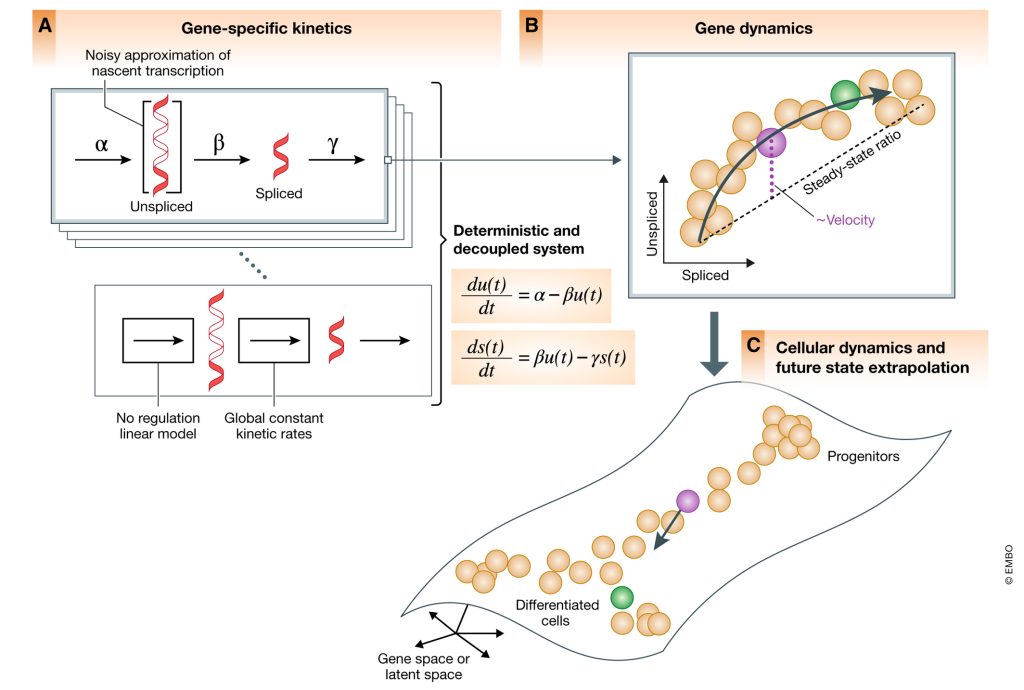
ただし、問題点も指摘されており、細胞を特徴づけるベクトルが遺伝子がごく限られた少数の遺伝子に従っているということが問題視されている(後述)。
このRNA velocityモデルについて、今回は2つのモデルを紹介する。velocyto と scVelo だ。
「定常状態モデル」velocyto
これは最初にRNA velocityが概念化されたときに作られたものである(La Manno et al., 2018)。
まず「定常状態」について触れておくと、mRNAのスプライシング速度、分解速度が時間経過によって変化しない状態、すなわち合成・分解速度が等しく、mRNA量が変化しない状態を指し示す。
例えば上のBの図でいうと、点線(Steady-state ratio)がそれに相当する。この状態よりも分解/生成が早かったり遅かったりすると細胞の状態、ベクトルが違ったものになっていく。
La Manno らは論文中ではほとんどの遺伝子の発現が定常状態、一定の傾きを持つ挙動を示すと述べている。一方で11%の遺伝子はそれとは異なる挙動を示し、組織特異的な選択的スプライシングもしくは分解速度を示唆していると述べている(La Manno et al., 2018)。
さて、それでは細胞の状態が変化する場合を考える。上の図に倣って転写速度を![]() 、スプライシング速度を
、スプライシング速度を![]() 、mRNAの分解速度を
、mRNAの分解速度を![]() としよう。
としよう。
遺伝子がアップレギュレートされている状態は、![]() が増加していると言い換えることができる。これによりスプライスされていない未成熟なmRNAが急速に増加し、スプライシング速度
が増加していると言い換えることができる。これによりスプライスされていない未成熟なmRNAが急速に増加し、スプライシング速度![]() に従って成熟mRNAとなり、それが定常状態に達するまで続く。
に従って成熟mRNAとなり、それが定常状態に達するまで続く。
逆にダウンレギュレートされている状態は、![]() の減少から始まり、スプライスされていない未成熟なmRNAの減少に引き続き、成熟mRNAの現象が引き起こされる。
の減少から始まり、スプライスされていない未成熟なmRNAの減少に引き続き、成熟mRNAの現象が引き起こされる。
つまり、スプライスされていない未成熟mRNAとスプライスされたmRNAの存在量の比というものによってこの細胞の「将来」の遺伝子発現の状態を知ることができるということだ。
この関係はスプライスされていない(![]() )、スプライスされた(
)、スプライスされた(![]() )mRNA量に基づき、以下の微分方程式によって表すことができる。
)mRNA量に基づき、以下の微分方程式によって表すことができる。
![]()
これは未成熟なmRNAと成熟mRNAの存在量の時間依存性(![]() )の関係を定量化するために、スプライスされたmRNA(
)の関係を定量化するために、スプライスされたmRNA(![]() )の存在量の一次微分を行っている。このモデルでは転写速度
)の存在量の一次微分を行っている。このモデルでは転写速度![]() が一定の場合、定常状態に漸近する。
が一定の場合、定常状態に漸近する。
![]()
このモデルはすべての細胞の均一な RNA velocity を仮定しているため、細胞部分集団のRNA velocityが類似していないような場合に予測性能が低下する可能性が指摘されている(Bergen et al., 2020; 2021; Li et al., 2024)。
「動的モデル」 scVelo
そこでこの定常状態の仮定を緩和したモデルが登場した。Bergen et al. (2020)にある scVelo だ。論文本文の言葉でいうと「尤度ベースの動的モデルを使用してスプライシング動態のすべての転写ダイナミクスを解くこと」でその仮定を緩和しているようだ。
velocyto が細胞全体の定常状態を仮定しているのに対し、このモデルでは遺伝子ごとの転写を考慮する。このモデルでは定常状態モデルとは対照的に、隣接する細胞間でより一貫した RNA velocity の推定値を出し、より転写状態を正確に捉えることができるようだ。
scVelo のインストールや利用方法はここから利用できる。
この scVelo のモデルはEMモデルとも呼ばれている(Gayoso et al., 2024; Li et al., 2024)。Gayoso らは論文中でこのEMモデルの問題点として、推定上の’driving’遺伝子をランク付けすることができるものの、遺伝子の尤度、視覚化、正確さとはあまり関係がないと挙げている。特に、このモデルで細胞のRNA velocityの視覚化は適切ではない場合があると指摘している。
まとめ
以上、RNA velocity の代表的(だと思われる)2つのモデルについて簡単に解説した。このRNA velocityの解析については今もなお発展が続いており、最後に紹介したGayosoらの2024年の論文ではRNA velocityを推定するための深層生成モデル(Deep Generative Model)、veloVI を開発している(Gayoso et al., 2024)。pip で入れられるので導入も簡単だが、ベンチマークしたほうがいいだろう。
かなりホットで活発な分野なので、これからまだまだ新しいモデルが開発されると思われるが、問題はscRNA-seqをする予算がどこにあるのかというところかな……。マウスなどをはじめとしたいわゆる「大衆向けモデル生物」では活発にシーケンスされ、そのデータも公開されているのでそれを再解析するなどの応用が進むだろう。
参考文献
- La Manno, G., Soldatov, R., Zeisel, A., Braun, E., Hochgerner, H., Petukhov, V., … & Kharchenko, P. V. (2018). RNA velocity of single cells. Nature, 560(7719), 494-498.
- Dvorak, P., Hanicinec, V., & Soucek, P. (2023). The position of the longest intron is related to biological functions in some human genes. Frontiers in Genetics, 13, 1085139.
- Bergen, V., Soldatov, R. A., Kharchenko, P. V., & Theis, F. J. (2021). RNA velocity—current challenges and future perspectives. Molecular systems biology, 17(8), e10282.
- Li, S., Zhang, P., Chen, W., Ye, L., Brannan, K. W., Le, N. T., … & Wang, G. (2024). A relay velocity model infers cell-dependent RNA velocity. Nature biotechnology, 42(1), 99-108.
- Bergen, V., Lange, M., Peidli, S., Wolf, F. A., & Theis, F. J. (2020). Generalizing RNA velocity to transient cell states through dynamical modeling. Nature biotechnology, 38(12), 1408-1414.
- Gayoso, A., Weiler, P., Lotfollahi, M., Klein, D., Hong, J., Streets, A., … & Yosef, N. (2024). Deep generative modeling of transcriptional dynamics for RNA velocity analysis in single cells. Nature methods, 21(1), 50-59.
- Li, J., Pan, X., Yuan, Y., & Shen, H. B. (2024). TFvelo: gene regulation inspired RNA velocity estimation. Nature Communications, 15(1), 1387.





![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



