トランスクリプトーム、とりわけRNA-seqはその組織で発現している遺伝子を定量したり、同定したり、あるいは何らかの条件によって発現量が変化するのを検出したりするのに便利な技術である。
最近は真核生物のゲノムが多く読まれるようになり、利用できるリソースは年々増えてきている。しかし、変わった生物を研究を人間にとっては必ずしも利用できるゲノムリソースがあるとは限らない。そうした研究対象を利用しながらも、トランスクリプトームで発現定量を行い、かつGene Ontology などの情報も利用してエンリッチメント解析やパスウェイ解析をしたいという研究者も多いだろう(私がそう)。
これまでそうした場合によく使われていたソフトウエアとしてはBLAST2GOというものがよく知られている。ただしこれは一部は無料であるが、シェアウェア(有料ソフト)である。他に同様な機能のソフトウエアはいくつか存在したが、フルでオープンソースというものは少なかった。
今回紹介する論文はENTAPというパイプラインソフトウエアである。今回の記事ではその論文の紹介にのみとどめ、次回の記事でその使い方を紹介する。
Hart, A. J., Ginzburg, S., Xu, M., Fisher, C. R., Rahmatpour, N., Mitton, J. B., … & Wegrzyn, J. L. (2020). EnTAP: bringing faster and smarter functional annotation to non‐model eukaryotic transcriptomes. Molecular Ecology Resources, 20(2), 591-604. リンク
背景
これまで、真核生物のゲノムは4,500種近くが読まれているが、染色体レベルのアセンブルは450未満、完全なゲノムは40未満にとどまっていた。この全ゲノムのリソースの不完全さによって、依然としてトランスクリプトームは注目され続けている。機能ゲノミクスのサブカテゴリに位置するTranscriptomicsは、コード領域の発現レベルの定量化に焦点を当てていた。
「RNA-seq」として知られるハイスループットRNAシーケンスは、ショートリードのシーケンスを利用してその発現量の違いを定量化する。良いリファレンスゲノムが存在する場合は、そのリファレンスゲノムに対してRNA-seqのシーケンスリードを貼り付ける(マッピング)することで定量化できるが、そのようなリソースが利用できないような生物種の場合、de novo アセンブルをする必要がある。de novo アセンブルの代表的なアセンブラとしてはTrinityが知られている。Trinity はショートリードの冗長部分を利用し、リード間でオーバーラップ(重なっている)部分を探す。それをもとにアセンブル(組み立て)を行うというような手法を取っている。ただ、それには当然ミスが生じ、キメラ配列であったり、本来と違って断片化された配列、間違ったContigsなどが生成されることがある。また、本来のリード数よりも3~4倍程度大きい転写産物が作られることにもつながっている。これはどれだけRNA抽出等を丁寧にやったとしても避けられない問題であるが、リファレンスゲノムが存在しない種にとっての唯一の手段であることには代わりはない。
RNA-seq解析においては、Differential expression gene (DEG; 発現変動遺伝子)の検出が行われることが多い。de novo アセンブルによって検出されたこれらの遺伝子はそのソフト・サンプル固有の識別子が割り当てられるが、これらにはアノテーション情報(遺伝子名やGOなど)は含まれないため、そこから直接的に解析に用いることは不可能である。ただ、これらのアノテーションの割当は、超面倒くさい。このアノテーションはそのアセンブリの結果と用いるパブリックデータベースの大きさ、すなわち情報量に左右される。これまでにこの工程を行うパイプラインとしては先に述べたBLAST2GO(Götz et al., 2008)や、Trinotate (https://github.com/Trinotate/Trinotate.github.io/wiki ) などがあり、配列類似性やタンパク質のドメイン推定、遺伝子ファミリー、Gene Ontology などの割当が可能である。ただこれらのパイプラインは使えるものの、実行時間が長く、数日から数週間を超えるものもある。また、これらは種を考慮しなかったり、ライブラリー汚染などを考慮しなかったりという問題点が存在した。
今回著者らはそれらの既存のパッケージの欠点を克服し、かつオープンソースのアノテーションパイプラインを構築した。ENTAP(Nは下付き文字)はDIAMONDというマルチデータベースに対して効率的な検索方法を実装したソフトウエアを組み込んでいる(Buchfink et al., 2015)。
また、従来までのパイプラインでは、アセンブルされたトランスクリプトームの中に汚染物質(例えば、対象の生物種以外の生物種のRNAなど)が含まれていることを考慮していなかった。ENTAPではこのことについても考慮がなされる。
Material and Methods
ENTAPを評価するために、3つの生物種のRNA-seqデータが用いられた。すべての生物はTrinity v2.06 (Funaria hygrometricaおよびPinus flexilis)、v2.2 (Entylia carinata)でアセンブルされた。
パブリックデータベースであるSwiss-Prot、NCBI RefSeq Complete、NCBI Taxonomy Database を利用し、汚染物質の評価・検出と対象データの分類学的関連性を評価した。
実行には、アセンブル済みのトランスクリプトームMulti FASTAファイルが必要なほか、最大5つのダイヤモンド形式のデータベースが必要である。今回はTrinityを使用してアセンブルしたものを使っているが、ENTAPは使用したアセンブラを認識したりしないため、アセンブラに関わらず使用することが可能である。その他、FPKM値を利用したフィルタリングを利用するためにSAMまたはBAM形式のインプットもできるらしいが今回は触れない。
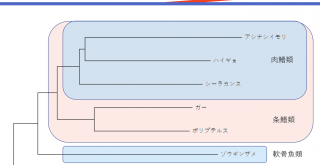
配列はDIAMONDデータベース内で評価され、またNCBI Taxonomy Database と比較することで汚染配列を削除する。遺伝子ファミリーの割当はEGGNOGデータベースを用いて行われる。GOの割当はGene Ontology データベースを用いて行われる。この辺はFig.1を参照。
結果
EnTAPを今回はTrinotateやBLAST2GOなどと比較した。
平均シーケンス長はフィルタリングの結果他よりも小さくなり、スプライスバリアントの存在などが減少した。
また、Trinotateと比較し、「より完全な遺伝子配列」が取れてくる割合が高くなることがわかった(Fig.2)。
肝心のアノテーションについてだが、ENTAPはSwiss-Prot/EGGNOGを用いたすべての種において、Trinotate、BLAST2GOと比較して全てにおいて最高のアノテーション率を得た。最低だったのがSwiss-Prot/PFamを用いたTrinotateであった。
また、Trianotateと比較して、そのアノテーションがどれほど一致するのかを検証した結果、例えばP. flexilis について、Trianotateによってのみアノテーションされたものが512個あったのに対し、EnTAPによってのみアノテーションされたものは4790個であった。この結果はEnTAPがEggNOGを採用しているのに対し、Trianotateは低品質なBLASTXに加え、HMMERを採用していることに起因していると述べている。
実行速度については明らかに他のソフトウエアよりも優秀で、例えばUniProt Swiss-Protの実行時間についてはF. hygrometrica についてはTrianotateで16時間、BLAST2GO PRO で82時間であるのに対し、EnTAPは1.1時間であった(Table 2)。NCBI RefSeq Complete についても明らかに他よりも実行速度が速かった。
あっさりまとめると、著者らの実験によればよく使われる2つのツールよりも明らかに今回のENTAPは高性能であるということだ。
参考文献リスト
- Hart, A. J., Ginzburg, S., Xu, M., Fisher, C. R., Rahmatpour, N., Mitton, J. B., … & Wegrzyn, J. L. (2020). EnTAP: bringing faster and smarter functional annotation to non‐model eukaryotic transcriptomes. Molecular Ecology Resources, 20(2), 591-604.
- Götz, S., García-Gómez, J. M., Terol, J., Williams, T. D., Nagaraj, S. H., Nueda, M. J., … & Conesa, A. (2008). High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic acids research, 36(10), 3420-3435.
- Buchfink, B., Xie, C., & Huson, D. H. (2015). Fast and sensitive protein alignment using DIAMOND. Nature methods, 12(1), 59-60.