今回は実践編。実際にデータを使ってやってみる。
EnTAP自体については前回の記事を参照して欲しい。
2020/07/20: 公式ドキュメントのリンクが変更されていたので貼り直した
動作環境
- Ubuntu 20.04 LTS
- Ryzen9 3900X (12コア24スレッド)
- RAM 64GB
- ストレージ 1TB SSD
正直ストレージが心もとないが、移行やらなんやらは面倒くさいので我慢。学振通ったら買いましょう。
基本的には公式ドキュメントを参照のこと。ただ微妙にソフトウエアが更新されたりとかあるので注意されたし。今回は公式ドキュメントよりも新しい実行コードを記述する。
依存ソフトのインストール
EnTAPに依存するためインストールが必要なソフトは以下の通り
- RSEM (Expression Filtering with alignment file): version 1.3.0
- GeneMarkS-T (Frame Selection): version 5.1
- DIAMOND (Similarity Search): version 0.9.9
- Version 0.8.31
- Version 0.9.19
- InterProScan (Protein Databases): This is not included within the EnTAP repo, but is supported
今回は基本的にすべて入っていないという前提で話をすすめる。
EnTAP本体のダウンロード
EnTAP本体はGitHubで公開されているのでそこからCloneしてくる。
$ git clone https://github.com/harta55/EnTAP.git
DIAMOND のインストール
DIAMONDは相同性検索に用いられるソフトウエアであり、今回のEnTAPの高速化に寄与していると言われているものでもある。
$ cd EnTAP/libs/diamond-0.9.9/ $ mkdir bin; cd bin $ cmake .. $ sudo make install $ make
もし正常にコンパイルされていたら、bin内で
$ ls CMakeCache.txt Makefile diamond CMakeFiles/ cmake_install.cmake install_manifest.txt
と出てくるはずである。
$ diamond help diamond v0.9.9.110 | by Benjamin Buchfink <buchfink@gmail.com> Licensed under the GNU AGPL <https://www.gnu.org/licenses/agpl.txt> Check http://github.com/bbuchfink/diamond for updates. (以下省略)
正常にコンパイルできた。
RSEM のインストール
わかりやすいようにカレントディレクトリも示しておく。先程のディレクトリからだとこのように移動すれば良い。もしEnTAPのrootディレクトリならcd libs/RSEM-1.3.0 で行ける。
kim@Ubuntu bin $ cd ../../ kim@Ubuntu libs $ cd RSEM-1.3.0/ kim@Ubuntu RSEM-1.3.0 $ make $ make ebseq $ sudo make install
GeneMarkS-T のダウンロード
アカデミック用途であればここからダウンロード可能。
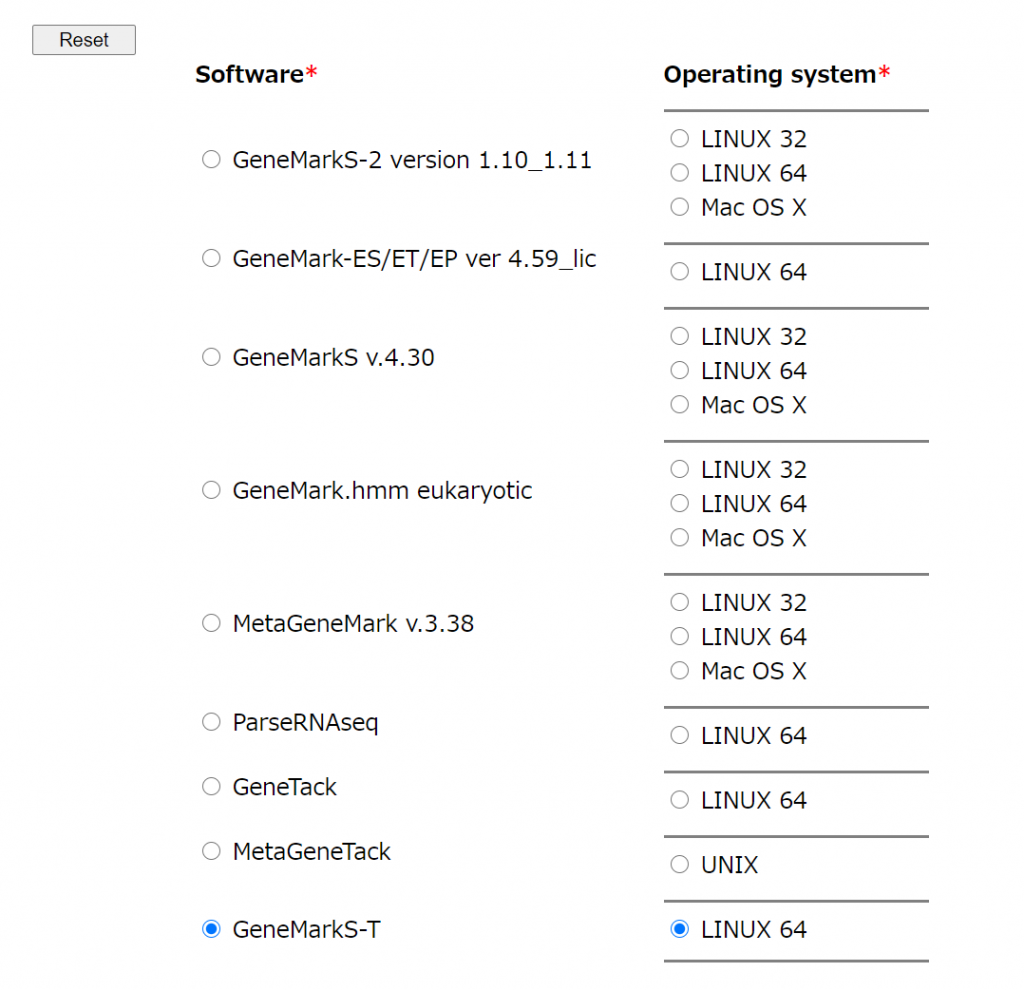
入力して契約に同意すると、ダウンロードリンクが出てくるので、ダウンロードしてやる。5MBくらい。
kim@Ubuntu RSEM-1.3.0 $ cd ../ kim@Ubuntu libs $ wget [ダウンロードリンク] $ mkdir gmst_linux_64 $ tar -zxvf gmst_linux_64.tar.gz -C gmst_linux_64/
ひとまずこれで依存ソフトウエアのインストールは完了である。
もし上記以外のディレクトリなどにインストールを行った場合は、別途そのパスをentap_config.txtに追記してやる。上記のパスの通りにインストールしていれば、その必要はない。
公式ドキュメントにはこのように書かれている。基本的に何も書かなければ以下のようなパスが与えられる。
- diamond_exe_path=/EnTAP/libs/diamond-0.9.9/bin/diamond
- rsem_exe_path=/EnTAP/libs/RSEM-1.3.0 (this is a path to the directory)
- genemarkst_exe_path=/EnTAP/libs/gmst_linux_64/gmst.pl
- eggnog_sql_database=/EnTAP/databases/eggnog.db (downloaded during Configuration)
- eggnog_dmnd_database=/EnTAP/bin/eggnog_proteins.dmnd (downloaded during Configuration)
- interpro_exe_path=interproscan.sh
- entap_database_sql_path=/EnTAP/databases/entap_database.db (text version, downloaded during Configuration)
- entap_database_bin_path=/EnTAP/bin/entap_database.bin (binary version, downloaded during Configuration)
- entap_graphing_script=/EnTAP/src/entap_graphing.py
ただ、私が行った環境ではデフォルトのパスではうまくいかなかったため、全て上記の/EnTAPを.に置換したものを用意した。
diamond_exe_path=./libs/diamond-0.9.9/bin/diamond rsem_exe_path=./libs/RSEM-1.3.0 genemarkst_exe_path=./libs/gmst_linux_64/gmst.pl eggnog_sql_database=./databases/eggnog.db eggnog_dmnd_database=./bin/eggnog_proteins.dmnd interpro_exe_path=./interproscan.sh entap_database_sql_path=./databases/entap_database.db entap_database_bin_path=./bin/entap_database.bin entap_graphing_script=./src/entap_graphing.py
EnTAP本体のインストール
最後にEnTAP本体のインストールを行う。EnTAPルートディレクトリに戻り、
$ cmake CMakeLists.txt $ make $ sudo make install
これでEnTAPのインストールが完了する。試しに$ EnTAP と入力すると実行可能であることがわかる。
データベースの準備
次に解析・アノテーションに使用するためのデータベースを準備する。これについては自分で好きなものを選んでくるほかないが、推奨されているものがいくつかあるのでその中から選ぶ。推奨されているデータベースはNCBIのRefSeq、Uniprot databases が挙げられている。
今回は対象の生物種が魚ということで、NCBI RefSeqの”other vertebrates”を使用することに加え、SwissProt と TrEMBL も使うこととした。少なくとも3つは選ぶと良いらしい。
まずデータベース保存用のディレクトリを適当なところに作った。今回はEnTAPのroot化にown_databaseを。
$ mkdir own_database; cd own_database
まずSwissProt と TrEMBL をダウンロード。
$ wget ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/complete/uniprot_sprot.fasta.gz $ wget ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/complete/uniprot_trembl.fasta.gz
TrEMBLが41GBもあったので寝た。寝て起きても終わってなかったが辛抱強く待ってチェックサムした。
また、RefSeqのOther_vertebratesからアミノ酸配列をダウンロードした。その後、解凍、統合。
kim@Ubuntu own_database $ mkdir vertebrate_other kim@Ubuntu own_database $ cd vertebrate_other/ kim@Ubuntu vertebrate_other $ wget ftp://ftp.ncbi.nlm.nih.gov/refseq/release/vertebrate_other/*.faa.gz $ gunzip -d * $ cat * > vertebrate_other.fasta
そして次にデータベースの構築を設定する。
$ EnTAP --config -d ./own_database/uniprot_sprot.fasta -d ./own_database/uniprot_trembl.fasta -d ./own_database/vertebrate_other/vertebrate_other.fasta --out-dir ./own_database/ -t 24
これでDIAMONDデータベースが構築された。
-t はスレッド数のオプションなので各自のCPUに合わせて指定する。自分のCPUのスレッド数より多くの数を指定すると問題が生じるのでコピペには注意。
テストデータでの検証
本番のデータに入る前にテストデータでの検証を行っておこう。
$ EnTAP --config -d ./test_data/swiss_prot_test.fasta --out-dir ./test_data
公式ドキュメントのものだとパスの仕様上Ubuntuではうまくいかないので注意。
次にテストデータでのパイプラインの実行を行いたいのだが、これもまた公式ドキュメントのコードデフォルトだと動かない。
entap_config.txt のバックアップを取った後、
diamond_exe_path=./libs/diamond-0.9.9/bin/diamond rsem_exe_path=./libs/RSEM-1.3.0 genemarkst_exe_path=./libs/gmst_linux_64/gmst.pl eggnog_sql_database=./test_data/databases/eggnog.db eggnog_dmnd_database=./test_data/bin/eggnog_proteins.dmnd interpro_exe_path=./interproscan.sh entap_database_sql_path=./test_data/databases/entap_database.db entap_database_bin_path=./test_data/bin/entap_database.bin entap_graphing_script=./src/entap_graphing.py
これに書き換える必要がある。
その後、’frame selection’ でのテストを走らせる。このフレームセレクションは転写産物のアセンブルされたコンティグにおいてコーディング領域がある場合のみそれを抽出するというものである。
$ EnTAP --runP -i ./test_data/trinity.fnn -d ./test_data/bin/swiss_prot_test.dmnd -t 24
フレームセレクションを行わない場合は以下のコマンド(今回は未検証)
$ EnTAP --runP -i ./test_data/trinity.faa -d ./test_data/swiss_prot_test.dmnd
それでしばらくすると、というかすぐに終わるので結果を確認する。
結果はカレントディレクトリ(EnTAP)の下の’entap_outfiles’に色々ディレクトリができている。必要に応じて中身を見る。例えば ‘final_results’ の中にはアノテーション済みのアミノ酸配列や塩基配列のMulti fasta ファイルが含まれている。
# 上記フレームセレクション有りでの出力例。final_annotated.faa >TRINITY_DN10071_c0_g1_i1len=618path=[1327:0-711328:72-951331:96-2361325:237-2601330:261-4361321:437-617][-1,1327,1328,1331,1325,1330,1321,-2] VKLPGPSIREPNVYDFGTPYRRMFDDLKRKDPELYKRNGLLQMLKRNLNVKMAPQRWQEN AVDGPFDVVFTFEERVFDMVIEDLQNREPIIMKSVLVVNLDVKDSHEEAAVGARLALDLC QKLEAVNAWEDEIDDIVMRFERQHRRKLIYTIYFY >TRINITY_DN10075_c0_g2_i1len=1094path=[2177:0-1062178:107-1402179:141-1093][-1,2177,2178,2179,-2] KGVSLWSLIKDNIGKDLTRVCLPVYFNEPISSLQKCFEDMEYSHLLDRAYEYGKQDNHLM RILHVAAFAVSGYASTAGRTCKPFNPLLGETYEADYPDKGLRFFSEKVSHHPMTIACHCR GRGWAFWGDSTLKSKFWGRSIQVDPVGILTVEFDDGEVFQWSKVTTTLYNLILGKINCDH YGVMHIKGNRLHSCKLKFKEQAIIERNPHQVQGYVHDRSGNKLATLVGKWDESMYFVMGD VALKSKSYDPMSGAMILWKKNDPPECPTRYNLTSFAITLNELTPGLKEKLPPTDSRLRPD QRHLECGEYDLANDEKMRLEQKQRQAYKLQEKGWKPRWFRRENDQSTYKYSGGYWEAREN ACWE >TRINITY_DN10075_c0_g1_i1len=1094path=[2180:0-1062181:107-1402182:141-1093][-1,2180,2181,2182,-2] KGVSLWSLIKDNIGKDLTRVCLPVYFNEPISSLQKYFEEMEYSHLLDRAYEYGKQDNHLM RILHVAAFAVSGYASTAGRTCKPFNPLLGETYEADYPDKGLRFFSEKVSHHPMTIACHCR GRGWAFWGDSTLKSKFWGRSIQVDPVGILTVEFDDGEVFQWSKVTTTLYNLILGKINCDH YGVMHIKGNRLHSCKLKFKEQAIIERNPHQVQGYVHDRSGNKLATLVGKWDESMYFVMGD VALKSKSYDPMSGAMILWKKNDPPECPTRYNLTSFAITLNELTPGLKEKLPPTDSRLRPD QRHLECGEYDLANDEKMRLEQKQRQAYKLQEKGWKPRWFRRENDQSTYKYSGGYWEAREN ACWE >TRINITY_DN10060_c0_g1_i1len=652path=[1293:0-4071294:408-4381295:439-651][-1,1293,1294,1295,-2] ERGVAGLYRGIGSNLASSAPISGIYTFTYESVKAALLPHLEKEYHAFAHCVAGGCASIAT SFIYTPSECVKQQMQVGSQYCNSWKALMGILEKGGFPLLYAGWGAVLCRNVPQSVIKFYT YEGLKHLALRRHSTEAHLGTLQTLAFGGLAGSTAALFTTPFDVIKTRLQTQILGSPTYYE GVFQAFQHIATHEGVGGLYRGLLPRLVIYISQGALFF >TRINITY_DN10010_c0_g1_i1len=1162path=[2421:0-4412422:442-4652423:466-4732436:474-6542435:655-9162431:917-9402432:941-1161][-1,2421,2422,2423,2436,2435,2431,2432,-2] VRKAGSKLARTGRVISPVAALPEALLFDCDGVLVDTERDGHRVSFNEAFSEKGLNVTWDV DLYGELLKIGGGKERMTAYFNKTGWPDIAPSTEGERKELIASLHRRKTQLFMALIEKRLL PLRPGVARLIDEALEKGVKVAICSTSNEKAVSAIVQCLLGPPRADAISIFAGDIVPHKKP DPAIYLLAATTLGVGTSRCVVIEDSAIGLAAAKAAGMKCIVTKSGYTVEEYFTSADAIFD DIGDPPNANFDLNFCGNLLEKQYAS
また、TSV形式(タブ区切りのフォーマット)でのアノテーション結果が test_data/final_annotations_test.tsv に保存されている。
TRINITY_DN10010_c0_g1_i1len=1162path=[2421:0-4412422:442-4652423:466-4732436:474-6542435:655-9162431:917-9402432:941-1161][-1,2421,2422,2423,2436,2435,2431,2432,-2] sp|Q94K71|CBBY_ARATH 65.7 303 95 2 28 324 20 319 3.7e-110 91.700000 sp|Q94K71|CBBY_ARATH CBBY-like protein OS=Arabidopsis thaliana GN=CBBY PE=1 SV=1 Arabidopsis thaliana /tempdata3/alex/thesis_testsets/limber_cuba/entap/8_threads/outfiles/similarity_search/blastp_Trinity_uniprot_sprot.out Complete No 39946.BGIOSGA012984-PA 5.6e-112 409.5 Viridiplantae 0TNAU@lilNOG,15J3S@poaNOG,1BF1D@strNOG,1DRP6@virNOG,COG0637@NOG,KOG2914@euNOG haloacid dehalogenase-like hydrolase family protein {"PFAM":[["Hydrolase",23,"100","100"]]} GO:0005575-cellular_component(L=0),GO:0005622-intracellular(L=3),GO:0005623-cell(L=1),GO:0005737-cytoplasm(L=4),GO:0009507-chloroplast(L=6),GO:0009526-plastid envelope(L=6),GO:0009532-plastid stroma(L=6),GO:0009536-plastid(L=5),GO:0009570-chloroplast stroma(L=7),GO:0009941-chloroplast envelope(L=7),GO:0031967-organelle envelope(L=3),GO:0031975-envelope(L=3),GO:0043226-organelle(L=1),GO:0043227-membrane-bounded organelle(L=2),GO:0043229-intracellular organelle(L=3),GO:0043231-intracellular membrane-bounded organelle(L=4),GO:0044422-organelle part(L=2),GO:0044424-intracellular part(L=3),GO:0044434-chloroplast part(L=6),GO:0044435-plastid part(L=5),GO:0044444-cytoplasmic part(L=4),GO:0044446-intracellular organelle part(L=3),GO:0044464-cell part(L=2),
TRINITY_DN10060_c0_g1_i1len=652path=[1293:0-4071294:408-4381295:439-651][-1,1293,1294,1295,-2] sp|Q94AG6|SAMC1_ARATH 37.0 216 123 4 6 217 95 301 5.8e-27 97.700000 sp|Q94AG6|SAMC1_ARATH S-adenosylmethionine carrier 1, chloroplastic/mitochondrial OS=Arabidopsis thaliana GN=SAMC1 PE=1 SV=1 Arabidopsis thaliana /tempdata3/alex/thesis_testsets/limber_cuba/entap/8_threads/outfiles/similarity_search/blastp_Trinity_uniprot_sprot.out Internal No 29760.VIT_02s0025g03490.t01 2.2e-83 313.9 Viridiplantae 10BII@NOG,1BIXB@strNOG,1DVBX@virNOG,KOG0768@euNOG Mitochondrial carrier protein {"PFAM":[["Mito_carr",66,"287","106"]]}
TRINITY_DN10075_c0_g1_i1len=1094path=[2180:0-1062181:107-1402182:141-1093][-1,2180,2181,2182,-2] sp|Q940Y1|ORP2A_ARATH 73.1 364 98 0 1 364 341 704 3.1e-166 100.000000 sp|Q940Y1|ORP2A_ARATH Oxysterol-binding protein-related protein 2A OS=Arabidopsis thaliana GN=ORP2A PE=2 SV=1 Arabidopsis thaliana /tempdata3/alex/thesis_testsets/limber_cuba/entap/8_threads/outfiles/similarity_search/blastp_Trinity_uniprot_sprot.out Internal No 4641.GSMUA_Achr10P19570_001 5.8e-174 615.5 Viridiplantae 0TU27@lilNOG,0XP9E@NOG,1BN54@strNOG,1DZ9R@virNOG,KOG1737@euNOG oxysterol-binding protein-related protein 1C-like {"PFAM":[["Oxysterol_BP",34,"100","100"],["PH",27,"79.4","98.7"]],"SMART":[["COIL",43,"126","101"],["PH",32,"94.1","99.7"],["TRANS",8,"23.5","92.1"]]}
TRINITY_DN10075_c0_g2_i1len=1094path=[2177:0-1062178:107-1402179:141-1093][-1,2177,2178,2179,-2] sp|Q940Y1|ORP2A_ARATH 73.6 364 96 0 1 364 341 704 2.5e-168 100.000000 sp|Q940Y1|ORP2A_ARATH Oxysterol-binding protein-related protein 2A OS=Arabidopsis thaliana GN=ORP2A PE=2 SV=1 Arabidopsis thaliana /tempdata3/alex/thesis_testsets/limber_cuba/entap/8_threads/outfiles/similarity_search/blastp_Trinity_uniprot_sprot.out Internal No 4641.GSMUA_Achr10P19570_001 4.8e-176 622.5 Viridiplantae 0TU27@lilNOG,0XP9E@NOG,1BN54@strNOG,1DZ9R@virNOG,KOG1737@euNOG oxysterol-binding protein-related protein 1C-like {"PFAM":[["Oxysterol_BP",34,"100","100"],["PH",27,"79.4","98.7"]],"SMART":[["COIL",43,"126","101"],["PH",32,"94.1","99.7"],["TRANS",8,"23.5","92.1"]]}
TRINITY_DN10071_c0_g1_i1len=618path=[1327:0-711328:72-951331:96-2361325:237-2601330:261-4361321:437-617][-1,1327,1328,1331,1325,1330,1321,-2] sp|Q558Z3|SSU72_DICDI 47.4 156 79 2 2 155 60 214 6.8e-38 99.400000 sp|Q558Z3|SSU72_DICDI RNA polymerase II subunit A C-terminal domain phosphatase SSU72 OS=Dictyostelium discoideum GN=ssu72 PE=3 SV=1 Dictyostelium discoideum /tempdata3/alex/thesis_testsets/limber_cuba/entap/8_threads/outfiles/similarity_search/blastp_Trinity_uniprot_sprot.out Partial 5 Prime No 29760.VIT_06s0009g01900.t01 3.4e-67 259.6 Viridiplantae 1BGFF@strNOG,1DQ89@virNOG,COG5211@NOG,KOG2424@euNOG RNA polymerase II subunit A C-terminal domain phosphatase [["mRNA surveillance pathway (03015)",15,"55.6","96.8"]] {"PFAM":[["Ssu72",27,"100","100"]],"SMART":[["COIL",2,"7.41","85.9"]]} GO:0005575-cellular_component(L=0),GO:0005622-intracellular(L=3),GO:0005623-cell(L=1),GO:0005737-cytoplasm(L=4),GO:0009507-chloroplast(L=6),GO:0009536-plastid(L=5),GO:0043226-organelle(L=1),GO:0043227-membrane-bounded organelle(L=2),GO:0043229-intracellular organelle(L=3),GO:0043231-intracellular membrane-bounded organelle(L=4),GO:0044424-intracellular part(L=3),GO:0044444-cytoplasmic part(L=4),GO:0044464-cell part(L=2),
ヨシ!
とりあえずテストデータでの実行が問題なければインストールやセットアップは問題ないであろう。
何かインストール等について質問や、間違っている所があれば上記ナビゲーションバーの「当サイトについて」のところからコメントを書き込んで伝えてください。



![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



