ラボの人に訊かれたのでついでにメモとして。私自身はシーケンスに出したりはしないが、そういうこともあるかもしれないので。
某ラボでは某クスのソフトを使ってシーケンス結果を見ているようだが、結構お高い有料ソフトなもので普通ならみんな導入できない。
今回はフリーソフトのみを使用して、ab1形式のシーケンスデータをFASTA形式のファイルに変換する。
一応 Windows / mac 対応
FinchTVのダウンロードとFASTAへの変換
まず最初にab1形式をFASTAに変換するソフト、FinchTVを導入する。
こちらからダウンロードできる。
インストールして、起動するとこんな画面が出てくる。
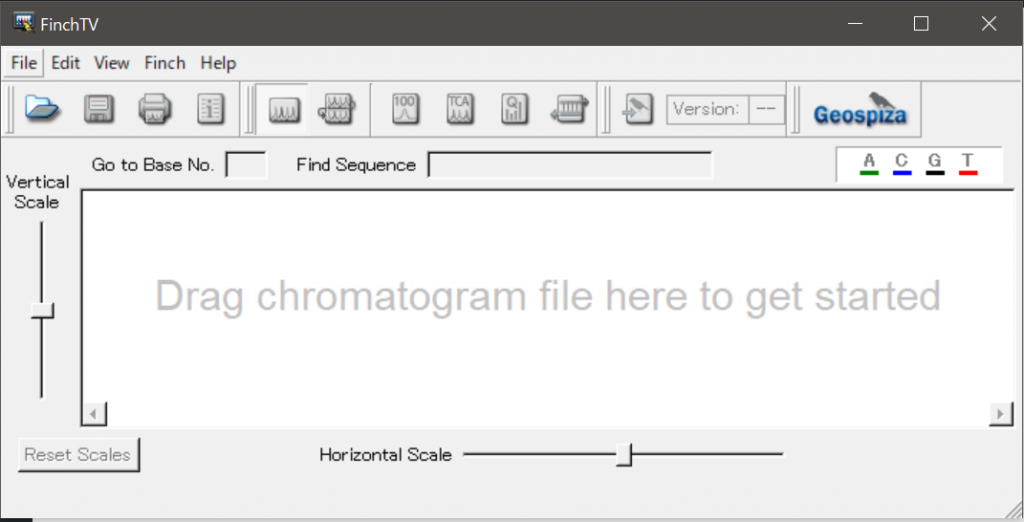
これにab1形式のファイルを突っ込んでやる
サンプルデータはこちらからお借りした。
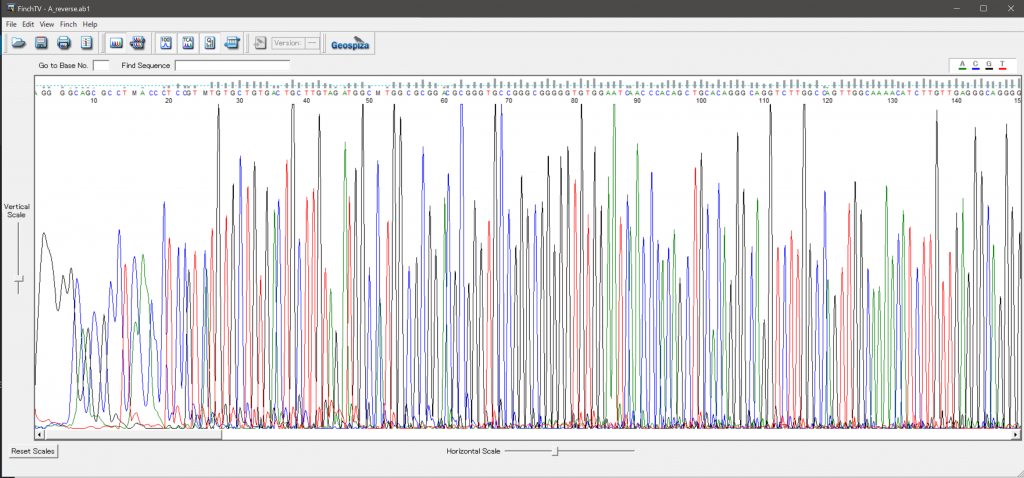
これでシーケンスの波形を見てやったりして状態を確認する。
これをFASTAファイルにするためには、File>Export>DNA sequence: FASTAをクリック。
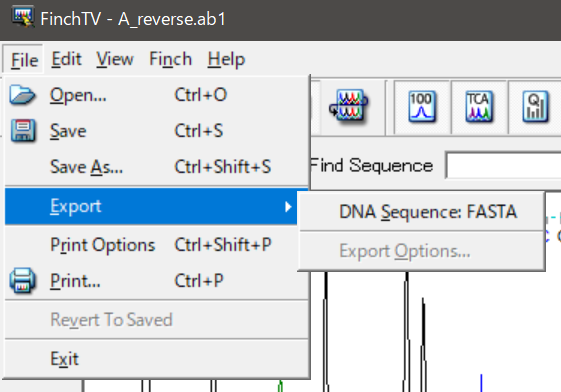
適当な場所に保存してやり、中身を見てやると

FASTA形式になっていることがわかる。先程もらったデータで実際にやってみたところ>の行が欠落している場合もあったので一応確認しておいたほうが良い。
今後の処理で少し躓く可能性があるので、ファイルの拡張子は’.fasta’にしておこう。
JalviewでFASTAファイルを開いてアライメント
インストールとファイルの読み込み
次にJalviewというソフトを使ってこのFASTAファイルを開き、アライメントしよう。
よく生物学の分野ではMEGAが用いられるが、大量のシーケンスとか読むときは正直重かったりするので私は使っていない。MEGAの記事は良く日本語でいっぱい書かれているのでそちらでやりたい方は検索しよう。
Jalviewはフリーソフトで、こちらからダウンロードできる。Javaが必要。
読み込み方はかんたんで、File > Input Alignment > From File
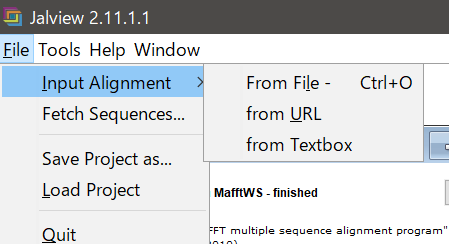
そして読み込みたいFASTAファイルを指定して開く。
今回は手間を省くために予め複数のFASTAファイルをまとめてMultiple fasta形式のファイルを1つ用意しておいた。1つずつ追加する方法ももちろん可能。
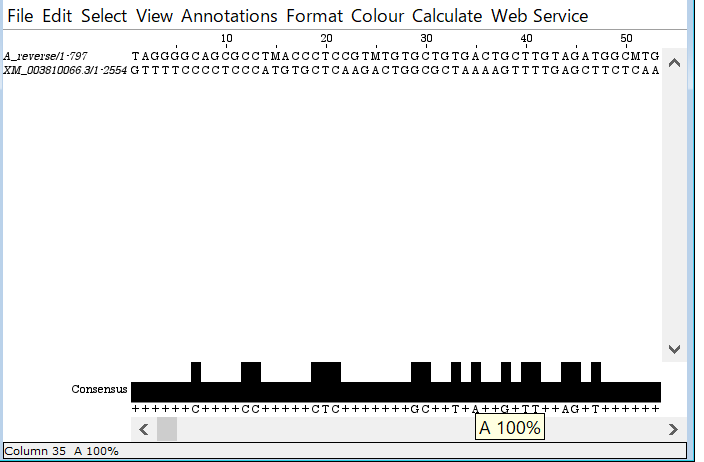
こんな感じのウィンドウが開く。デフォルトのままだと塩基が判別しづらいので私はいつもカラーを付けている。
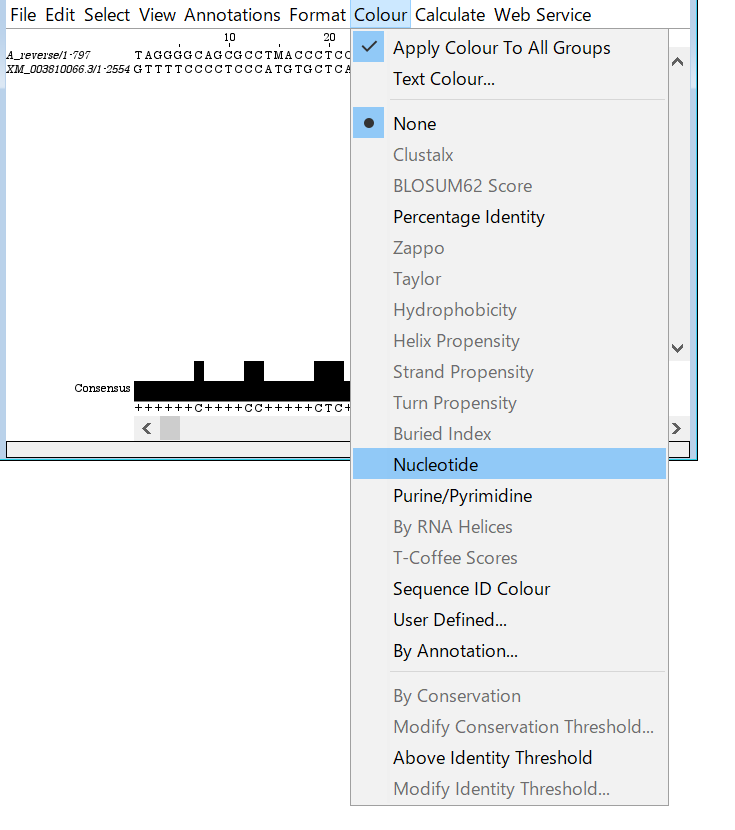
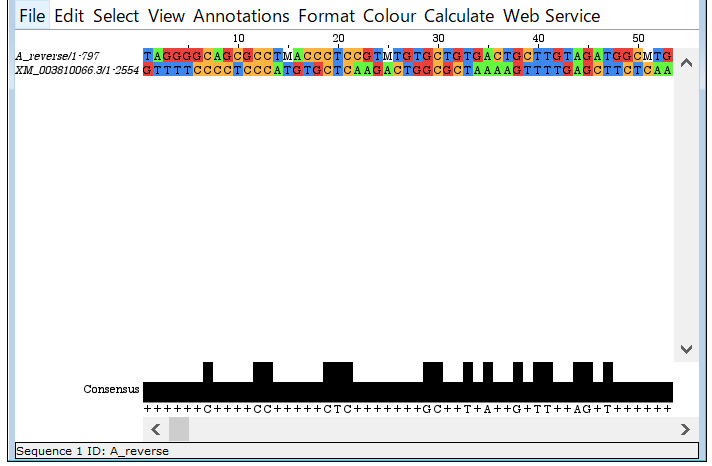
色がついた。
アミノ酸配列でも可能で、色々と色を付けることができる。
アライメント
さて、最後にアライメントをしよう。JalviewはMEGAと違ってローカルリソースを使わず、Webサーバを経由して様々な機能を提供している。簡単に言えば非力なパソコンでも解析が可能ということである。
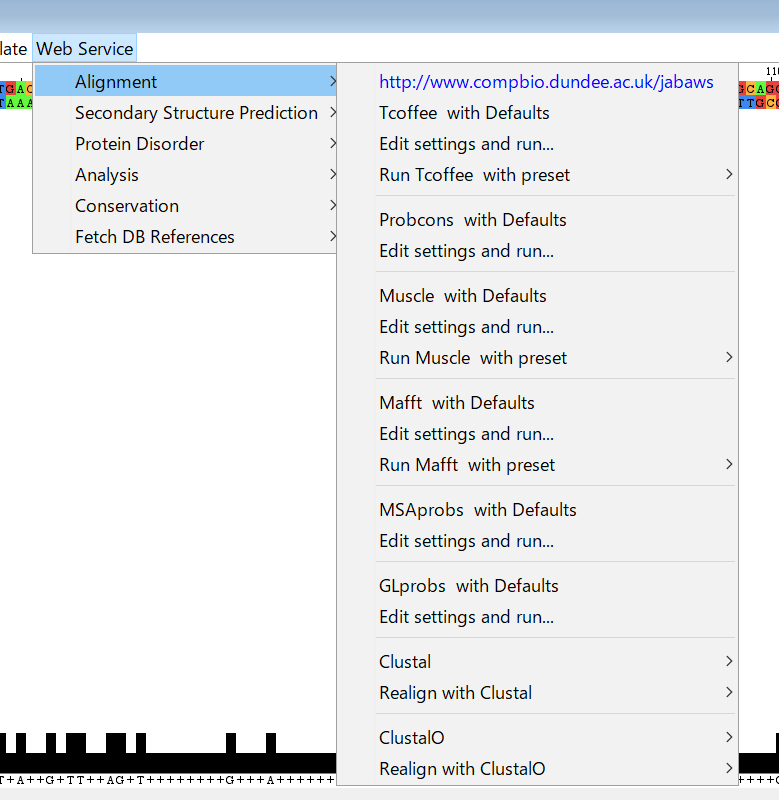
Web service > Alignment からお好みのアライメントソフトウエアを選択。実行。今回は私お気に入りの軽量・高速アライメントツールであるMAFFTを選択。
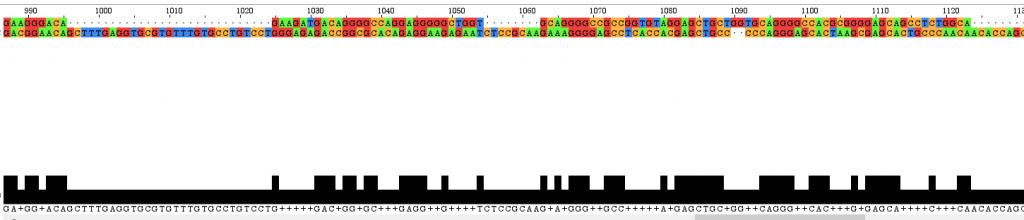
今回はテキトーに選んだシーケンスなので正解はよくわからんが、アライメントがきちんとされる。
おわりに
今回はab1ファイルからFASTA形式への変換、Jalviewでの確認とアライメントの手法を軽く説明した。
どれもフリーソフトなのでお金のないラボや有料ソフトアレルギーの人間でも安心して使える。お金がないからってライセンス違反行為をするのは、やめましょう。
おしまい。


![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



