転移因子(トランスポゾン、TE)はゲノムのある位置からある位置へと飛んでいくというDNA配列である。様々な真核生物においてゲノムに存在し、様々な役割を担っていたり、あるいは単純に「ジャンクDNA」であるようなものも存在する(最初はジャンクとして見られていたけど近年になって様々な役割が明らかにされてきているという感じ)。
ゲノムサイズを増加させる要因にもなっており、ハイギョや有尾両生類においてはその巨大ゲノムに寄与している(Wang et al., 2021)。哺乳類においては25-50%ほどがTEであるとも言われている(Platt et al., 2018)。
TEにも色々と種類があり、分類がなされている(Wicker et al., 2007)。このウィッカーの分類法に基づくと、TEはまずクラスIとクラスIIに分類される。クラスIはレトロトランスポゾンとも呼ばれ、転置にRNAの中間体を経由してDNAに入り込む。さらにレトロトランスポゾン(クラスIのTE)は更に大きく5つに分類でき、
- 長い反復配列であるLTR
- チロシンリコンビナーゼ(YR)をコードするDIRS、Ngaro、VIPERなどを含むDIRS (Dictyostelium Intermediate Repeat Sequences)。※ ただし哺乳類ゲノムにはDIRSはないことが知られている(Piednoël et al., 2011)。
- Penelope-like Elements (PLE)
- LINE
- SINE
などに分けられる。細かい解説も書こうと思ったが長くなりそうなので別記事にまとめることにした。
また、クラスIIのTE(カットアンドペーストで、RNAを中間体としない)には、
- Subclass I
- TIRS
- Crypton
- Subclass II
- Helitron
- Maverick
などの分類が存在する。
転移因子(TE)はゲノム中に多く存在するため、上記のように階層的、体系的に分類していくことが必要となる。
これに対して、分類法も色々と開発されてきている。これまでの多くのツールは隠れマルコフモデル(HMM)を使用したものや、既知のTEとの相同性、機械学習(ML)を用いた分類などであった。TEclass、REPCLASS、PASTEC、LTRclassifier等々。
今回の記事では機械学習を用いたトランスポゾン分類法について最近の2例を取り上げて紹介したい。
ClassifyTE
今回紹介する1つ目は転移因子(TE)を階層的に分類するためのスタッキングベースの機械学習フレームワークであるClassifyTEである(Panta et al., 2021)。
ClassifyTEは様々な機械学習による分類のパフォーマンスを調べた上で実装されたものである。
実際に使ってみたが、簡単に使用することはできた。GitHubでソースコードが公開されているため、環境さえあれば簡単に実行できる。
しかし問題は微妙な精度だ。
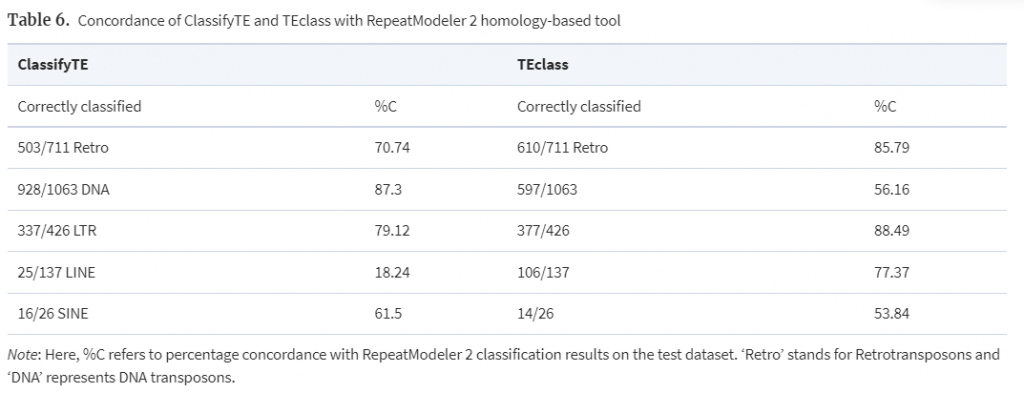
人気のTE分類ソフトであるTEclassと比較したものだが、DNA以外ではほとんど劣っていると言って良い。特にLINEの分類は致命的なまでに低い。RepeatModeler2 (Flynn et al., 2020) で分類したものを正解とした場合、という前提があるためもちろんその前提が間違っている可能性も無きにしもあらずだが、相同性ベースのため信頼性はそちらに軍配があがるだろう。
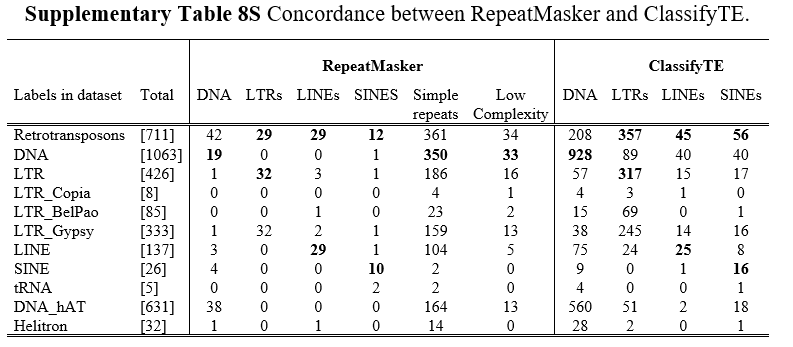
ただRepeatMaskerと比較した際はより良好な分類結果を示していると見てよいだろう。LINEは劣っているが。
このLINEの精度が悪い原因としては学習に用いたデータベースにLINEの数が少なかったということらしい。
DeepTE
他の機械学習を用いた最近のツールにDeepTE (Yan et al., 2020)がある。文字通り深層学習(ディープラーニング)が使用されている。最近のゲノム科学、進化生物学の分野にも機械学習の波がやってきているのがBioinformaticsなどのジャーナルを眺めているとなんとなく感じる。
これは先程のClassifyTEとは違うモデルなのだが、検証が別の方法を取っているので両者を正当に比べることができない。一応パフォーマンスについてはSupplement Dataで掲載されているが(下図)、本文中では先の論文とは異なる人気分類ツール、PASTECと比較していた。
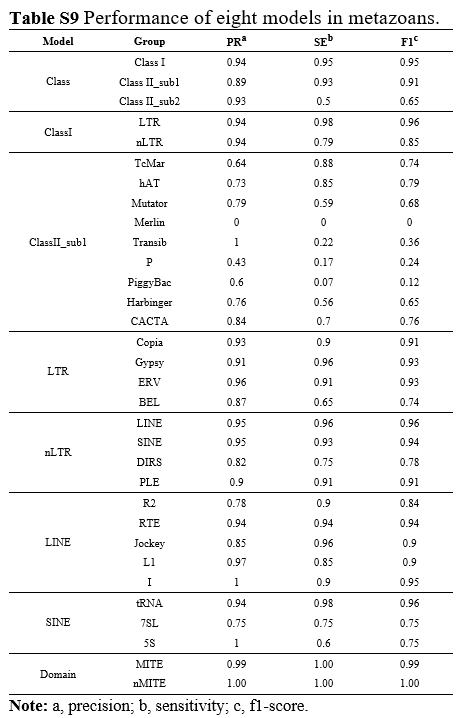
植物ゲノムのグループが作っているようだが、ちゃんと後生動物にも適応可能であると示している。
こちらもGitHubでコードが公開されているので利用可能だ。
この論文のイントロでは指摘されているが、RepeatModelerは既知のデータベースをもととした検出をしているため、未知のTEに弱いという面もある。そうしたことからdenovoのTE配列を探る上ではこうしたツールを利用するというのも大いに有効ではないだろうか。
一応、現在脊椎動物最大のゲノムのハイギョ(オーストラリアハイギョ)の論文(Meyer et al., 2021)では、RepeatModelerなどに加え、このDeepTEとPASTECが両方用いられている。
参考文献
- Panta, M., Mishra, A., Hoque, M. T., & Atallah, J. (2021). ClassifyTE: a stacking-based prediction of hierarchical classification of transposable elements. Bioinformatics.
- Yan, H., Bombarely, A., & Li, S. (2020). DeepTE: a computational method for de novo classification of transposons with convolutional neural network. Bioinformatics, 36(15), 4269-4275.
- Wang, K., Wang, J., Zhu, C., Yang, L., Ren, Y., Ruan, J., … & Wang, W. Cell 184.5 (2021): 1362-1376.
- Platt, R. N., Vandewege, M. W., & Ray, D. A. (2018). Mammalian transposable elements and their impacts on genome evolution. Chromosome Research, 26(1), 25-43.
- Wicker, T., Sabot, F., Hua-Van, A., Bennetzen, J. L., Capy, P., Chalhoub, B., … & Schulman, A. H. (2007). A unified classification system for eukaryotic transposable elements. Nature Reviews Genetics, 8(12), 973-982.
- Piednoël, M., Gonçalves, I. R., Higuet, D., & Bonnivard, E. (2011). Eukaryote DIRS1-like retrotransposons: an overview. BMC genomics, 12(1), 1-18.
- Flynn, J. M., Hubley, R., Goubert, C., Rosen, J., Clark, A. G., Feschotte, C., & Smit, A. F. (2020). RepeatModeler2 for automated genomic discovery of transposable element families. Proceedings of the National Academy of Sciences, 117(17), 9451-9457.
- Meyer, A., Schloissnig, S., Franchini, P., Du, K., Woltering, J. M., Irisarri, I., … & Schartl, M. (2021). Giant lungfish genome elucidates the conquest of land by vertebrates. Nature, 590(7845), 284-289.



![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



