今回論文を作成するにあたってggplot2を用いて有意差を示すアスタリスクとバーを含むきれいなグラフを作りたい、ということから勉強して作成した。
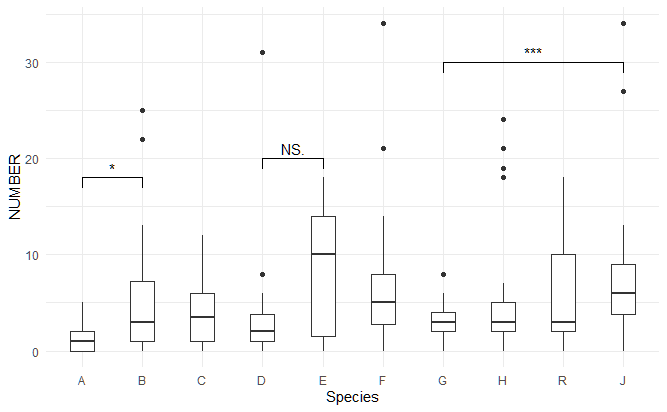
出来上がった図はこちら。
前回はライブラリ(ggplot2)を使わずに作成したが、以下のようなものだった。
ダサかったね。現時点で3000アクセス以上あったけど、ごめんなさい。
RとR studioをインストール
RとR studioをインストールしておこう。特にR studioはR notebookを使って「ノートを取るように」解析結果を残しておくことができる。コマンドライン上でちまちまやっていると、その実行したコマンドが記録に残らなかったりしてあとから図の修正を行う際に面倒なことになる。
スクリプトファイルにしておくというのも手だが、いちいち図を出力し直したりするのが面倒くさい。
今回はRのバージョン4以上を対象に行っていく。
必要なライブラリのインストール
library(ggplot2)
library(ggsignif)でエラー無く実行できるなら既にインストールされているので問題ない。もしインストールされていないのであれば以下のコマンドを入力してインストールする必要がある
install.packages("ggplot2")
install.packages("ggsignif")これで1つ上のコマンドを実行すれば、問題なく実行できるはずである(何も表示されない)
インストールは完了。
ggplot2 で箱ひげ図なり棒グラフなりを作ろう
まずは自分の作りたい図をイメージしよう。
先の図は箱ひげ図(Box plot)であったが、場合によっては棒グラフなどを使うこともあるだろう。
ある生物種ごとにケラチンに含まれるアミノ酸残基数をカウントし、それをプロットしてやりたかった。与えてやるデータセットは以下のような形式である。
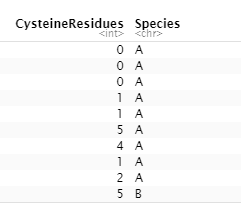
raw_data <- read.table(file='sample.txt', sep='\t', header = T, fileEncoding="UTF-8")
num <- transform(raw_data, Species= factor(Species, levels = c('A', 'B', 'C', 'D', 'E', 'F', 'G', 'H', 'R', 'J')))
library(ggplot2)
plt <- ggplot(num, aes(y=CysteineResidues,x=Species)) +
geom_boxplot(width=0.4) +
ylab("NUMBER") + expand_limits(y = 0)
pltこのようにすると、
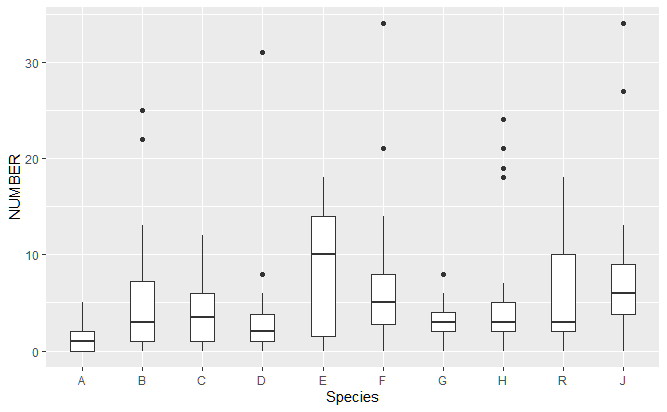
こんな感じのものがプロットされる。これだけでもなんとなく見栄えのする図となる。ggplot2スゲー。こだわる人間はもっと色々調節していくがそれは後ほど。
箱ひげ図の読み方については今私と同年代の人間は高校の数学Aだったかでやったはずなので教科書を見直そう。
検定を行って有意差のアスタリスクと”バー”をつける
次にこれらの種間(A-J)の間で数に統計的に有意な差が存在するかを明らかにすべく、検定を行う。通常、Rではデフォルトでt検定などの検定関数が入っているため、検定だけならば追加のライブラリは必要ないものが多い。
今回はこのggplot2のグラフに重ねる形でアスタリスクやバーを追加したいので、ggsignifというライブラリを使う。
使い方は非常にかんたん。
library("ggsignif")
plt2 <- plt + geom_signif(comparisons = list(c("A", "B")),
test = "t.test",
na.rm = FALSE,
map_signif_level = TRUE,
y_position = 18,
col = "red") + theme_minimal()
plt2これによって以下の図が得られる。
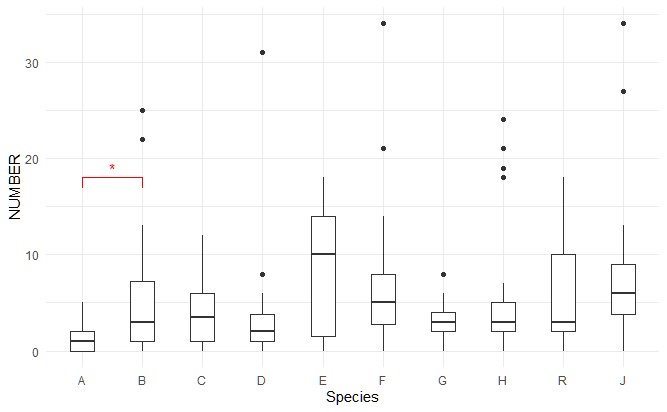
わかりやすいように赤で示しているが、見た目が悪いので通常は黒に近い色にしたほうがいいだろう。
ggsignifの詳細
comparisons
comparisons = list(c("A", "B"))の部分で、比べる群を指定している。リスト形式にして渡す必要がある。
test
test = "t.test"で行う検定の種類を選んでいる。今回はt-test(t検定)を指定した。ただし、ここでt検定を用いることは正しくないかもしれないことに注意すること。t検定は2群ともに分布が正規分布に従っているかを検定した上でとか色々前提条件があったりする。他にはwilcox.testなどを指定することも可能だ。こちらはノンパラメトリック用の検定となっている。
na.rm
na.rm = FALSEはデフォルトでFALSEである。FALSEならば欠落データがあった場合に警告してくれる。Trueにすると警告なしに実行される。
map_signif_level
map_signif_levelはTRUEにすると上記のようにアスタリスクつき(p<0.05で*、p<0.01 **, p<0.001 ***)の表示にしてくれる。FALSEにすると以下のようにp値をバーの上に表示する形式になる。
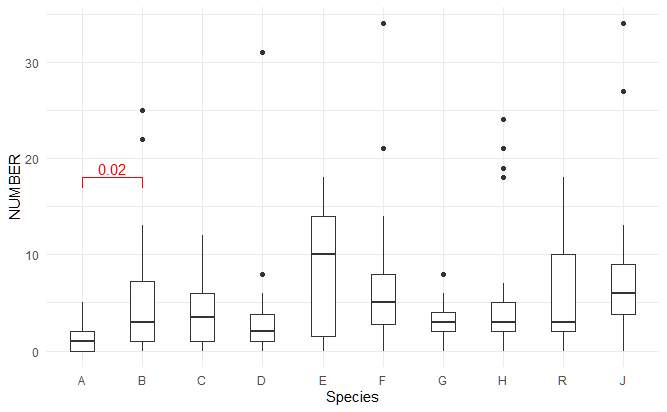
また、マニュアルでアスタリスクとp値の関係を変えることができる。例えばもっと低いp値の記述をアスタリスク4個でしたい場合は以下のようになる。
plt2 <- plt + geom_signif(comparisons = list(c("A", "B")),
test = "t.test",
na.rm = FALSE,
map_signif_level = c("****" = 0.0001, "***" = 0.001, "**" = 0.01, "*" = 0.05),
y_position = 18,
col = "red") + theme_minimal()分野によってこのアスタリスクとp値の関係が違ったりするものだが、必ず論文にはアスタリスクの数とp値の関係を示す必要がある。自明だと思わないこと。
y_position
y_positionによって、そのバーの場所を変えることができる。
y軸の値に対応しているので、一旦出力してどこに描画するかを調整しつつやっていくのが良さそうである。
col
col = "red"の部分を修正することでバーやアスタリスク、p値の色を変えられる。Rに登録されている色の単語の他に、カラーコードで指定してやることもできる。
***
今回はさらにtheme_minimal()を追加し、ggplot2によって標準出力されるグラフの見た目を変更した。こっちのほうがスッキリしていて見やすい。これは好みだと思う。
“+”でつないでいき、複数これを繰り返していくことでいくらでも2群間の検定をすることができる。
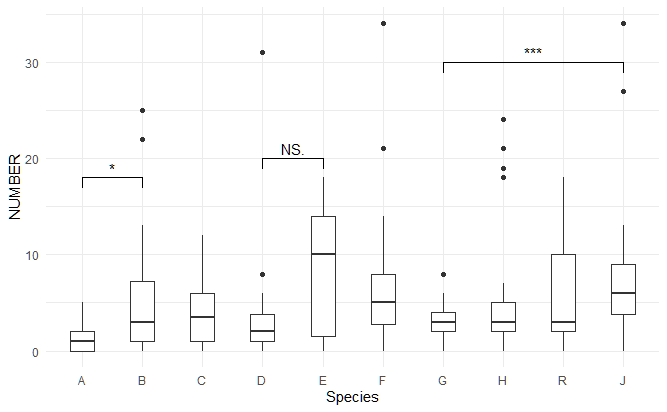
きれいにかけてうれしいね。
おわり。





![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)


