2005年の愛・地球博でユカギルマンモスの展示を見たことがある。「ただ毛が生えたゾウだなあ」くらいにしか思わなかったが、マンモスは現在地球上に一頭も残っていない「過去の生物」である。
多くの文献では人間の狩猟によってその個体数を減らし、絶滅したと考えられている。しかし Wrangel 島にかつて生息していたマンモスはそうではなかった可能性が最新の研究から明らかにされた。
今回は進化生物学の分野で著名な雑誌である”Genome Biology and Evolution” (通称GBE)に2020年3月に掲載された論文を紹介する。
Erin Fry, Sun K Kim, Sravanthi Chigurapti, Katelyn M Mika, Aakrosh Ratan, Alexander Dammermann, Brian J Mitchell, Webb Miller, Vincent J Lynch, Functional Architecture of Deleterious Genetic Variants in the Genome of a Wrangel Island Mammoth, Genome Biology and Evolution, Volume 12, Issue 3, March 2020, Pages 48–58, https://doi.org/10.1093/gbe/evz279

これまでの通説
マンモスはいつまでいたのか
本土(ユーラシア大陸)のマンモスは10,500年前ごろに絶滅したと言われている。ただ、それより前に海面上昇によりセントポール島とウランゲリ島にマンモスが取り残されたことが知られている (Vartanyan et al., 2008)。
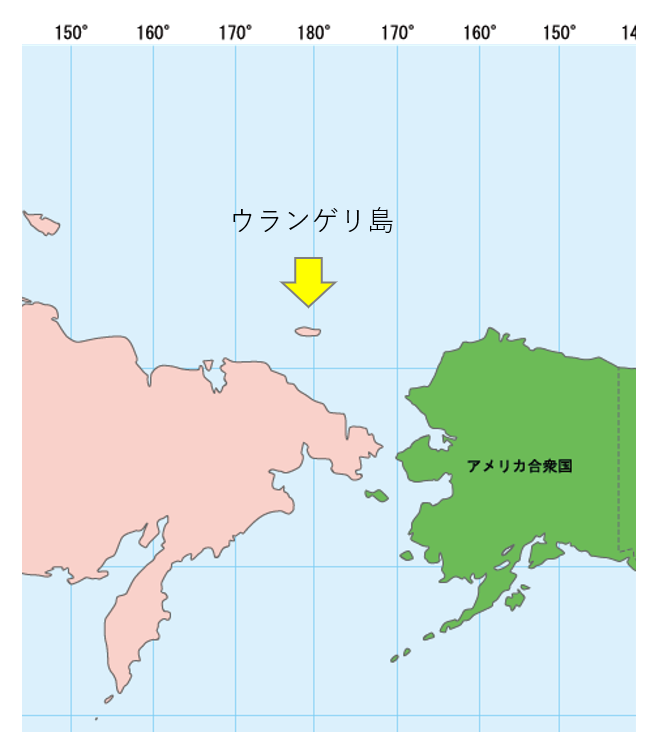
本土のほうのマンモスについては生息地の消失と人間の狩猟の組み合わせによって絶滅した可能性が言われているが(例えば Lorenzen et al., 2011)、島にはまだ人間が移入していなかったことが指摘されている。本土から隔離されたこれら島のマンモスについては島面積の減少が絶滅の要因となっているとされてきた。
なぜ島の面積が減少することが絶滅に繋がるのか?
島面積の減少というものはすなわちマンモスの生息できる環境(食べ物など)を制限するというものである。これによってマンモスの集団の個体数が減少するという事象が起きるのだが、集団の個体数が一定以上を超えて減少するとどんどん「遺伝的多様性」が失われていく。つまるところ、近親交配のようなことが繰り返し起こるようになっていくことでゲノムに蓄積された有害な変異が集団の中で固定されていくという自体につながっていく。
有害な変異はすぐさまマンモスに死をもたらすようなものではなくとも徐々に「集団としての適応度」を低めていくものとなる。
これに関して詳しく知りたい方は以下の記事を参照していただきたい。
今回使用するマンモスのDNA
今回解析に使用されたマンモスのゲノムDNAは過去2015年に発表されたものを使用している(Palkopoulou et al., 2015)。簡単にその論文を紹介しておくと、ロシアの北東部シベリアあたりの氷漬けのマンモスおよびウランゲリ島のマンモスからDNAを抽出して全ゲノムシーケンス(おおよそすべてのDNAの配列を決定すること)を行ったという内容である。
これらのDNAの断片はすべてアフリカゾウのゲノムに対してマッピング(塩基配列同士をコンピュータ上で貼り付けること)を行い、比較がしやすいように遺伝子の位置や向きなどの整理を行った。
また著者らは今回有害な変異について「ホモ接合」であるものに限って調べることにした。理由は単純で、長い年月によるDNAの損傷によって本来あったはずではない変異も検知してしまう可能性があるからである。ヘテロ接合の可能性のものを排除し、ホモ接合で変異があるものを選び出すことで有害なアミノ酸置換を検出したようだ。
また今回有害変異が確認された遺伝子について、マウスやツメガエルを使った組み換え実験を行っているが、本記事では割愛する。
結果
ウランゲリ島のマンモスのゲノムを他のマンモスゲノムのものと比較し、一塩基置換の変異を洗い出し、そこから非同義置換(塩基配列の変異によりコードされるアミノ酸配列が変わってしまうような変異のこと)のものを抽出した。 Polyphen-2 というソフトウエアとマウスのノックアウト変異体の表現型を用いて、ウランゲリ島マンモスの112の遺伝子について115の「有害の可能性が高い」非同義置換があったことを同定した。
一体どんな遺伝子に変異があったのか?
ではその「有害な変異」があった遺伝子とはどのようなものであったのだろうか。
具体的に少し列挙すると Hydrolethalus Syndrome Protein 1(HYLS1) などが上げられる。これは繊毛形成に関与することが知られているタンパク質をコードする遺伝子であり、これが異常となると水頭症などの重度な脳の奇形や、正中線の欠如などをもたらす。つまりこのマンモスが属していた集団では高い頻度でそうした脳の発生に異常が生じていた可能性が示唆された。
また、ウランゲリ島マンモスゲノムに関して以前から嗅覚受容体に関する遺伝子の偽遺伝子化(コードされるアミノ酸が機能しないこと)が指摘されていた。今回有害な変異が115個見つかったわけだが、その中でも21個が嗅覚受容体に関わるものであった。
例えばOR5A1という遺伝子に変異が見つかっているが、これはβイオノンという匂いを受容するタンパク質をコードしている。βイオノンはバラやスミレなどの香りの主成分であり、ウランゲリ島マンモスではこれが壊れているという。つまり、花の匂いをかげなかった可能性があるのだ。ただ、もしかしたら当時生息していた花の組成がこのマンモスの好みに関わる遺伝子に影響を与えていたのかもしれない。つまるところ、βイオノンを発するような花を好んで食ったりしなくなった可能性もあるということだ。

どうしてウランゲリ島マンモスは絶滅したか
一般的に野生個体群の遺伝的多様性を維持するためには最小でも500個体必要であるという。ただ有害な変異の蓄積を避けるためには最低1,000個体は必要だと言われている(Nunney and Campbell., 1993)。例えばアジアゾウだと218~4,700と見積もられている。そしてこのウランゲリ島マンモスの個体数は300~500個体ほどであったと推定されている。これは島の生息可能個体数の理論値である150~800個体に近い。
今回のゲノム解析の結果を踏まえると、ウランゲリ島マンモスは集団の個体数が小さいため、有害な突然変異を排除することができなかったことが考えられる。
結局今回の研究からはどの変異が原因というところまではわからなかった。ただこれは永久にわからないだろうし、おそらく複合的な要因が複雑に絡み合って起きた結果絶滅した可能性が高い。
今回の研究からわかったこととしては、もうウランゲリ島マンモスの集団が絶滅しようとしているとき、その集団に属していたマンモスはどんな病気や障害を抱えていたのかということだ。
以前にも遺伝的多様性についての記事を書いたことがあるが、一度失われた遺伝的多様性を取り戻すのは難しい。もしいま人間が愛する動物の個体数が激減しているのならば、このマンモスたちのような運命を回避する方法を取るべきであろう。
関連: なぜ「失われた生き物をよみがえらせる」プロジェクトはダメだと言われるのか
参考文献リスト
- Erin Fry, Sun K Kim, Sravanthi Chigurapti, Katelyn M Mika, Aakrosh Ratan, Alexander Dammermann, Brian J Mitchell, Webb Miller, Vincent J Lynch, Functional Architecture of Deleterious Genetic Variants in the Genome of a Wrangel Island Mammoth, Genome Biology and Evolution, Volume 12, Issue 3, March 2020, Pages 48–58, https://doi.org/10.1093/gbe/evz279
- Vartanyan, S. L., Arslanov, K. A., Karhu, J. A., Possnert, G., & Sulerzhitsky, L. D. (2008). Collection of radiocarbon dates on the mammoths (Mammuthus primigenius) and other genera of Wrangel Island, northeast Siberia, Russia. Quaternary Research, 70(1), 51-59.
- Lorenzen, E. D., Nogués-Bravo, D., Orlando, L., Weinstock, J., Binladen, J., Marske, K. A., … & Ho, S. Y. (2011). Species-specific responses of Late Quaternary megafauna to climate and humans. Nature, 479(7373), 359-364.
- Palkopoulou, E., Mallick, S., Skoglund, P., Enk, J., Rohland, N., Li, H., … & Reich, D. (2015). Complete genomes reveal signatures of demographic and genetic declines in the woolly mammoth. Current Biology, 25(10), 1395-1400.
- Nunney, L., & Campbell, K. A. (1993). Assessing minimum viable population size: demography meets population genetics. Trends in Ecology & Evolution, 8(7), 234-239.


![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



