今回紹介するのはRaNA-seqというWebサービスだ。
Carlos Prieto, David Barrios, RaNA-Seq: interactive RNA-Seq analysis from FASTQ files to functional analysis, Bioinformatics, , btz854, https://doi.org/10.1093/bioinformatics/btz854
RaNA-seq に使われている技術
RNA-seqの解析にはこれまで度々記事にしてきたが、結構なマシンパワーとそれなりのコスト・技術・知識が必要となる。
しかし近年開発された「マッピング・カウントをしない」タイプのトランスクリプト定量化手法である “Salmon” (Patro et al., 2017) は非常に低コストで発現遺伝子の定量化が可能である。
ざっくりここで手法を述べておくと、全トランスクリプトームの配列情報、アノテーションをインデックス化し、それに対してアライメントを行い、定量化する。
同様にマッピングを行わない手法としては kallisto がよく知られているが、 Salmon のほうが偽陽性が少ないとかなんとか。
RaNA-seq にはこの Salmon が使用されており、高速に解析が可能というわけである。
どんな感じ?
こらちにアクセスしてみて欲しい。 https://ranaseq.eu/
会員登録が必要だが、基本無料のようだ。
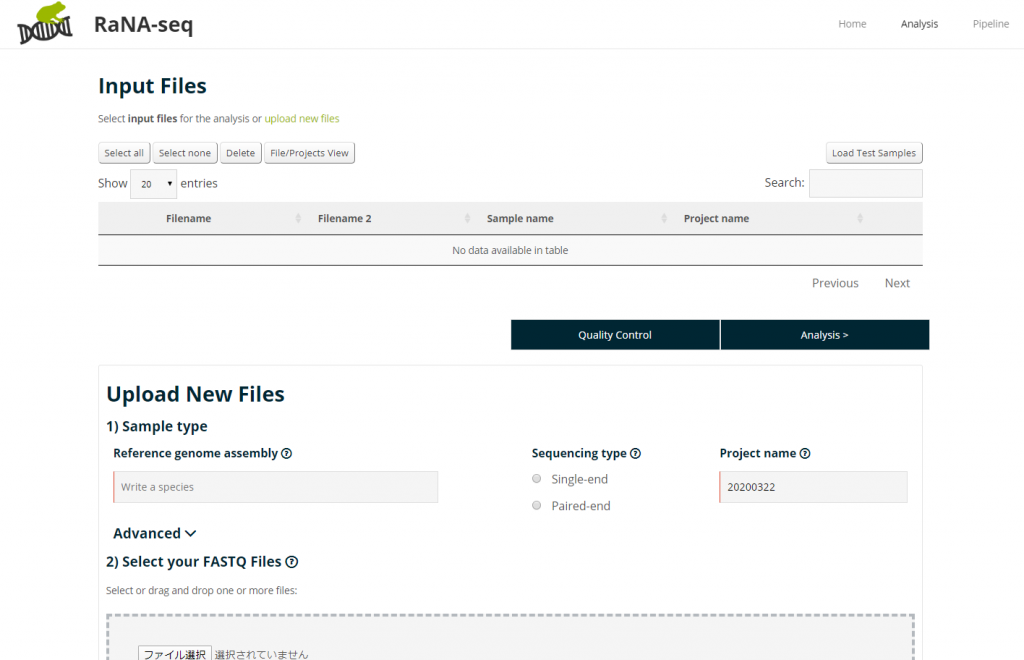
本当にただFastqファイルをアップロードするだけで解析ができる模様。
定量化は TPM、解析結果の可視化はもちろんのこと、GO との連携もしているようで、Over presented analysis や、Geneset enriched などの解析ツールが用意されている模様。すべてWeb上で完結する。
参考文献リスト
- Carlos Prieto, David Barrios, RaNA-Seq: interactive RNA-Seq analysis from FASTQ files to functional analysis, Bioinformatics, , btz854, https://doi.org/10.1093/bioinformatics/btz854
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., & Kingsford, C. (2017). Salmon provides fast and bias-aware quantification of transcript expression. Nature methods, 14(4), 417.


![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



