NCBI、よく使うのだがIDの重複がデータベース間で存在していたり、そもそもGeneIDを用いたデータベースをまたいだ検索ができないためストレスフルだった。数個ならいいのだが、今回は数十個あってさすがに1つ1つポチポチ手作業でやるのはバカのやることなのでツールを作ることにした。
NCBI Geneの検索結果から入手できるTabularフォーマットをもとにFASTAファイルを一括自動ダウンロードできる。対応は、mRNA、CDS、およびアミノ酸配列である。スプライシングバリアントにも対応しており、1つのIDから複数のスプライシングバリアントの配列を取ってくることが可能だ。
以下のGitHubページにおいて公開している。
https://github.com/kim2039/NCBI_geneIDtoFASTA
使い方等は(拙い)英語、日本語で書かれているのでリンク先を参照してほしい。
NCBIのAPIでFASTA取得を自動化したい
最初に疑問だったのが、そもそもこれを自動化することが可能なのであろうかといたことであった。正直NCBIのResultsから得られるSummaryなどは形式がズタズタで利用が非常にしにくい。今回は正規表現を使うことでなんとか取ってこれたが、いかんせんヒトが読みやすいが再利用がしにくい形式ばかりである。
それとFASTAを自動取得するにはどうしたらいいかといったことが問題になった。
ただ実は古くよりNCBIには Entrez Programming Utilities なるものが存在し、自動的にデータベースから配列をダウンロードすることが可能であったようだ。大学の学部の頃いちいち1つ1つページに飛んで数回の操作のあとにファイルをダウンロードして……ってことを繰り返すことを教わったがそんな事する必要ない、APIを使ってこちらで自動的に取得してやればいいのだ。
ただ日本語の記事は皆無に等しく、公式ドキュメントを読む他なかったが、今回は配列の取得に限ってメモしておく。
コードを見ていただければわかるが、配列の取得は以下のようなリンクを使っている。
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=nuccore&id=123456&rettype=fasta&retmode=text
idのところにそのデータベースに応じたIDを入れてやればFASTAが手に入るというものだ。そのデータベースに応じたIDというのがキモで、例えば上記のはNucleotideのデータベースを参照しているため、GeneIDを入れてやっても正しく参照してくれない。困ったちゃんだ。
ちなみにこれのidはNucleotideデータベースのIDを入れてやればよいのだが、今回はCDSやアミノ酸配列の取得も視野に入れている。いちいちそれぞれ取得するの心底面倒くさいと思っていたが、ドキュメントのところにCDSとアミノ酸配列を直接このNucleotideデータベースから取得できるとのことだったので、今回はmRNAのIDの取得を自動化するだけで済んだ。
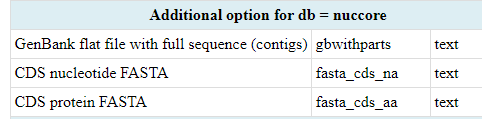
引用元: Entrez Programming Utilities Help [Internet].
他にもいろいろなオプションが存在しているので様々な自動化ツールを作ることができそうだ。
あとはこれをウェブスクレイピングの要領で取得してやって、出力して……ってことを繰り返してやれば良い。Pythonで書かれているので興味があれば見てください。
あ~~~~~楽になった。



![【Qiskit】マルチオミクス解析を量子機械学習でやる①[環境構築・基礎]](https://kimbio.info/wp-content/uploads/2024/05/2203027-100x100.jpg)



